
C'est le cas des gènes V et J des locus IGK, IGL, TRA et TRG.
Exemple: la séquence réarrangée de numéro d'accès AB006843
Lorque l'on lit les annotations IMGT (utilisation de IMGT/LIGM-DB), on remarque que cette séquence réarrangée de chaîne légère kappa est issue du réarrangement des gènes et allèles: IGKV2-28*01 [ou IGKV2D-28*01 (score identique)] et IGKJ2*01.
Si l'on examine dans IMGT Repertoire > Alignments of alleles, les gènes V et J germline dont la séquence est issue, on observe:
C'est le cas des gènes V, D et J des locus IGH, TRB et TRD.
Exemple: la séquence réarrangée de numéro d'accès AF052379
Lorque l'on lit les annotations IMGT (utilisation de IMGT/LIGM-DB), on remarque que cette séquence réarrangée de chaîne lourde est issue du réarrangement des gènes et allèles: IGHV5-51*03, IGHD1-7*01 et IGHJ4*02.
Si l'on examine dans IMGT Repertoire > Alignments of alleles, les gènes V, D et J germline dont la séquence est issue, on observe:
Non, il n'est pas facile de repérer le gène D dans une jonction V-D-J. En effet, c'est un gène court (quelques dizaines de nucléotides seulement) qui subit des mutations somatiques ainsi que des délétions et des insertions de nucléotides (N-diversité) à ses extrémités 5' et 3'.
Exemple de résultats:
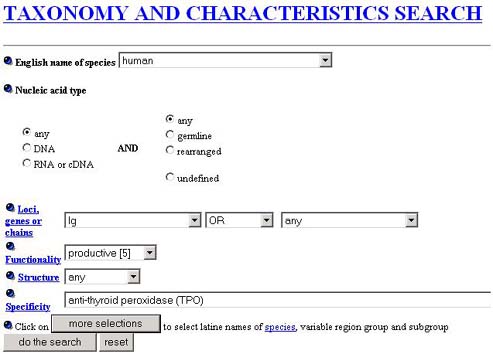
268 séquences (à la date du 01/07/2003)
Exemple de résultats:
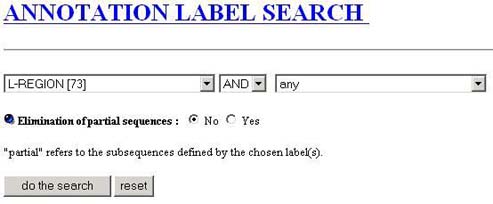
22 séquences (à la date du 01/07/2003)
| ID: | IDentification line |
| AC: | ACcession number line |
| DT: | DaTe line |
| OS: | Organism Species |
| OC: | Organism Classification |
| DE: | DEscription line |
| KW: | KeyWord lines |
| FT: | Feature Table lines |
| R -: | RN: Reference Number, RC: Reference Comment, RP: Reference Positions, RX: Reference Cross-reference, RA: Reference Author(s), RT: Reference Title, RL: Reference Location |
| SQ: | SeQuence header |
IGHV1-69*06
IGHD3-10*01
IGHJ6*01
Exemple : la séquence de numéro d'accès M11954
Au niveau d'IMGT/LIGM-DB, l'option "9 IMGT/V-QUEST analysis" permet d'obtenir la traduction de la JUNCTION comme suit :
Translation of the JUNCTION
104 118
C S A K L V G T D T Q Y F G P G
M11954 TGC AGT GCT AAA CTA GTA GGT ACA GAT ACG CAG TAT TTT GGC CCA GGC
>M11954,TRBV20-1*03,TRBJ2-3*01
TGC AGT GCT AAA CTA GTA GGT ACA GAT ACG CAG TAT TTT
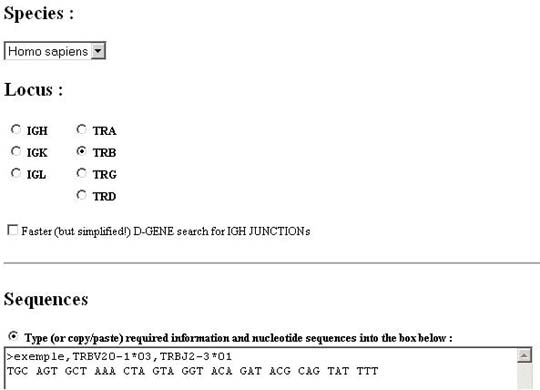
Dans notre exemple:

Ngc=2/6, cela signifie que parmi les 6 nucléotides ajoutés lors de la N-diversité, 2 sont des C ou G.
Oui, il s'agit d'une adénine (A) en 5' de la V-REGION.
Le CDR3 a une longueur de 11 acides aminés.
En effet, la Cys 104 (2nd-CYS) et le Trp/Phe 118 (J-TRP/J-PHE) ne sont pas inclus dans le CDR3-IMGT.
Par contre, ils font partie de la jonction et en marquent les extrémités.
→ Voir les réponses
← Masquer les réponses